Эпигенетика
Что такое эпигенетика?
Эпигенетика это наука об изменениях в экспрессии генов, вызванных механизмами, не затрагивающими последовательность ДНК. Но что именно это означает?
Многоклеточные организмы (как вы!) содержат одну и ту же последовательность ДНК во всех ядрах всех типов клеток, но при этом клетки кожи можно точно отличить от гепатоцитов или кардиомиоцитов. Механизмы, лежащие в основе создания этих специфических типов клеток, были названы «эпигенетической» регуляцией.
.png)
Греческий префикс «эпи-» означает «на» или «над» последовательностью ДНК. После десятилетий исследований этих эпигенетических модификаций или «маркеров», на сегодня эпигенетика обозначает наследственные модификации гистонов и метилирование ДНК, которые могут влиять на активность гена, без изменений основной последовательности ДНК.
Область эпигенетической регуляции значительно расширилась. Модификации гистонов (такие как ацетилирование и метилирование остатков лизина в коровых белках) служат для деконденсации или, наоборот, конденсации («упаковки») хроматина в «открытые» или «закрытые» конфигурации, связанные с «включенной» или «выключенной» экспрессией гена. Экспрессия некоторых некодирующих РНК помогает регулировать экспрессию генов во время или после транскрипции. Метилирование ДНК - еще одна ключевая модификация, при которой метильные группы добавляются к цитозинам, расположенным в сайтах цитозин-фосфат-гуанин (CpG) по всему геному.
Какова функция эпигенетической модификации?
Необходимость различных модификаций заключается в скоординированной регуляции экспрессии генов. Исследования показывают, что эти маркеры служат молекулярным механизмом, который опосредует влияние окружающей среды на геном, начиная с эмбрионального развития. Например, образ жизни и условия окружающей среды матери на ранних сроках беременности могут иметь долгосрочное влияние на здоровье ее потомства.1 Сообщается, что на метилом ДНК новорожденных влияет курение матери.2 Существуют доказательства, что некоторые эпигенетические метки передаются по наследству из поколения в поколение, несмотря на то, что большая часть этих маркеров впервые возникает на самом раннем этапе эмбриогенеза. При рождении и с течением времени, по мере того как мы стареем, паттерны метилирования ДНК продолжают меняться. Недавние исследования также показали, что уровни метилирования в определенных локусах можно использовать в качестве индикатора биологического возраста человека.3
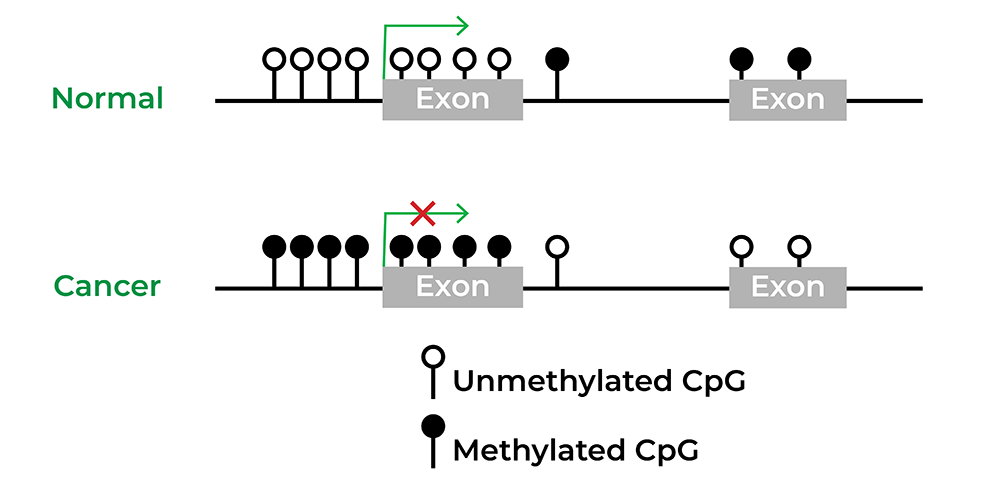
Эпигенетика также широко используется в лечении заболеваний, особенно рака. Несколько проектов, таких как Cancer Genome Atlas или TCGA, были разработаны для получения эпигеномов заболеваний человека. С помощью этих крупных когортных исследований были обнаружены и даже коммерциализированны биомаркеры для различных видов рака, включая рак толстой кишки и рак мочевого пузыря.4 Эпигенетические препараты доказали свою эффективность для лечения гематологических и плотных опухолей. В настоящее время проводится множество клинических испытаний, направленных на разработку низкомолекулярных молекул-ингибиторов, которые нацелены на изменение эпигенома рака отдельно или в сочетании с другой терапией.5
Как изучать эпигенетические изменения?
Наиболее изученным эпигенетическим маркером является метилирование ДНК. Стандартные методы секвенирования не работают для прямого анализа метилирования ДНК, потому что метильная группа ковалентно связана с цитозином (5-метилцитозин; 5-mС). Конверсия ДНК бисульфитом считается золотым стандартом для выявления изменений в 5-mС. Этот химический процесс дезаминирует цитозины и превращает их в урацил, если метильные группы не присутствуют в молекулах цитозина. При использовании секвенирования следующего поколения (NGS) метилированные цитозины читаются в последовательности как «C», а цитозины, конвертированные в урацил, читаются как «T». Сравнивая эти последовательности с референсным геномом, можно рассчитать процент метилирования с разрешением до одного основания. Бисульфитное секвенирование ограниченного набора геномных локусов (RRBS), полногеномное бисульфитное секвенирование (WGBS) и таргетное бисульфитное секвенирование являются наиболее широко распространенными технологиями NGS для определения профиля метилома.
С другой стороны существуют методы не применяющие бисульфитную конверсию, такие как иммунопреципитация метилированной ДНК (MeDIP). Они основаны на использовании антител, распознающих 5-mС, применяемых для обогащения метилированной ДНК из пула фрагментированной геномной ДНК. Эту обогащенную фракцию затем можно использовать для анализа метилирования всего генома. Возможно также высокопроизводительное количественное определение 5-метилцитозина (5-mC) в ДНК из различных образцов, используя те же антитела для связывания 5-mC в сочетании с технологией ИФА.
Как анализировать структурные изменения хроматина
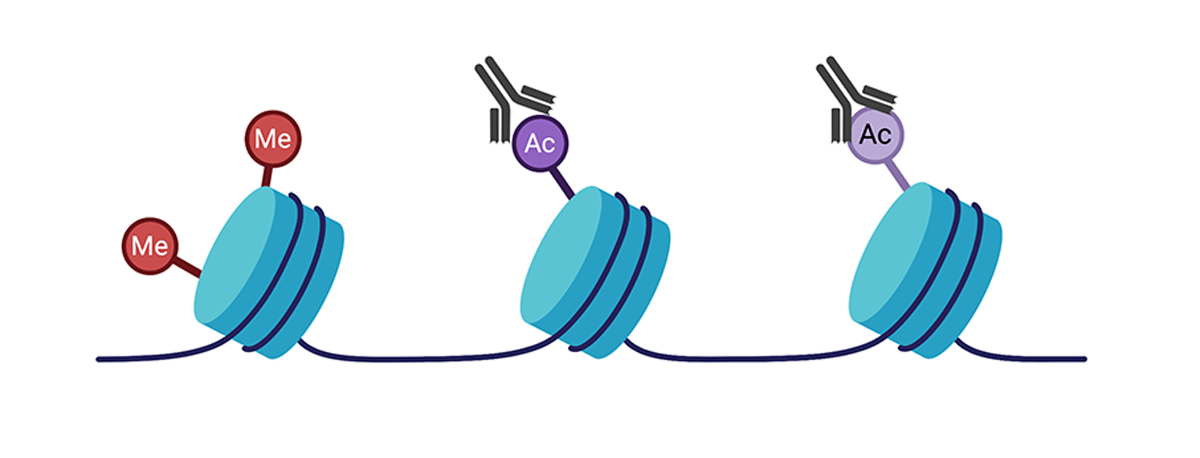
Модификации гистонов представляют собой ковалентные модификации коровых белков гистонов, которые могут изменять экспрессию генов. С помощью перекрестного связывания белков с ДНК можно использовать специфические для гистоновых белков антитела и селективно выделить связанные фрагменты ДНК. Этот метод называется иммунопреципитацией хроматина (или ChIP). Затем обогащенные фрагменты ДНК можно секвенировать (ChIP-seq) с помощью NGS для получения полногеномного профиля конкретной модификации гистона. Методы ChIP-seq также можно использовать для анализа факторов транскрипции и других сайтов связывания ферментов в геноме.
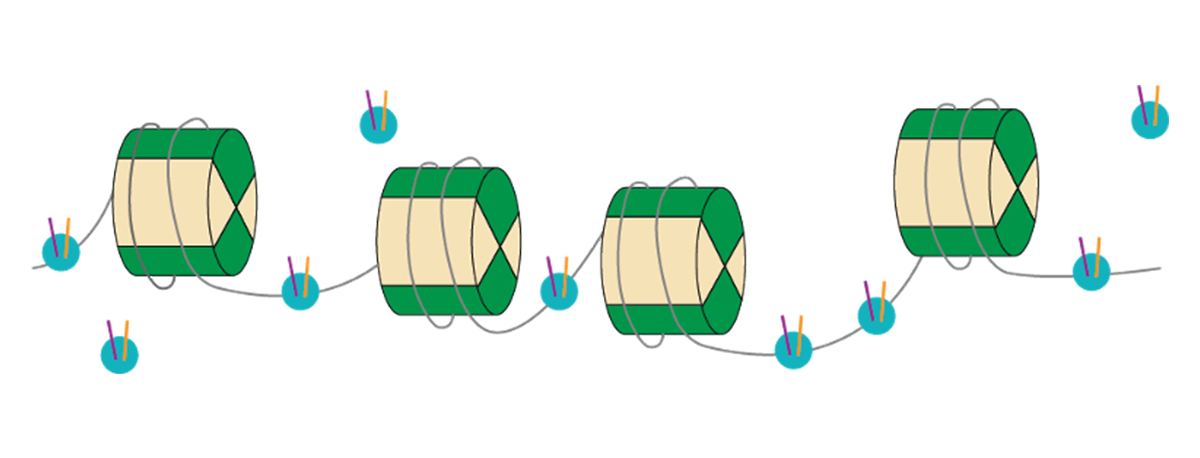
Поскольку доступность хроматина в его «открытой» конфигурации обычно связана с экспрессией генов, методы, которые определяют наличие или отсутствие неконденсированного хроматина, помогают нам понять важность изменений структуры хроматина в эпигенетическом ландшафте. Наиболее часто используемый метод определения доступности хроматина - это ATAC-seq (или анализ хроматина, доступного для транспозаз). Он позволяет оценить доступность ДНК. Этот мощный метод основан на способности фермента транспозазы вставлять адаптерные последовательности только в «открытые» сайты хроматина. При «более глубоком» секвенировании можно также различать уникальные сайты связывания или следы факторов транскрипции, которые могут связываться внутри нативного хроматина.
Будущее эпигенетических исследований
Благодаря сочетанию всех вышеперечисленных методов с секвенированием РНК можно напрямую оценить влияние эпигенетических изменений с использованием «многомерных» подходов. Например, объединяя информацию о маркерах метилирования, доступности хроматина и экспрессии генов можно выявить отдельные механизмы, на которые оказывает влияние окружающая среда в долгосрочных исследованиях (например, анализ повторного отбора проб крови у одного и того же человека)6.
Еще более информативным является совместное применение этих методов в исследовании единичных клеток. В связи с недавним быстрым увеличением мощности секвенирования в сочетании со значительным снижением затрат, резко возрос интерес к исследованию эпигеномики отдельных клеток для создания карт, специфичных для разных типов клеток. Проект Human Cell Atlas, возглавляемый рядом выдающихся ученых в этой области, стремится создать исчерпывающие справочные карты всех человеческих клеток. Это откроет новые уровни понимания клеточного развития, идентичности и функционирования при болезни. Например, полногеномное бисульфитное секвенирование (WGBS), проведенное для единичных клеток печени мыши, выявило удивительно высокий уровень гетерогенности внутри ткани печени.7 Поскольку цены на секвенирование продолжают падать и разрабатываются новые методы, появятся еще более захватывающие открытия в области эпигенетики.
Продукция для эпигенетики
Методы с использованием бисульфита | |
Бисульфитная конверсия | |
NGS -исследования | Pico Methyl-Seq Library Prep Kit |
ПЦР-методики | |
Методы без использования бисульфита | |
Методы на основе антител | |
Энзиматические методы | |
Анализ хроматина | |
Преципитация хроматина ChIP | |
Структура хроматина | |
Литература:
1. Toraño, E. G.; García, M. G.; Fernández-Morera, J. L.; Niño-García, P.; Fernández, A. F., The Impact of External Factors on the Epigenome: In Utero and over Lifetime. Biomed Res Int 2016, 2016, 2568635.
2. Joubert, B. R.; Felix, J. F.; Yousefi, P.; Bakulski, K. M.; Just, A. C.; Breton, C.; Reese, S. E.; Markunas, C. A.; Richmond, R. C.; Xu, C. J.; Küpers, L. K.; Oh, S. S.; Hoyo, C.; Gruzieva, O.; Söderhäll, C.; Salas, L. A.; Baïz, N.; Zhang, H.; Lepeule, J.; Ruiz, C.; Ligthart, S.; Wang, T.; Taylor, J. A.; Duijts, L.; Sharp, G. C.; Jankipersadsing, S. A.; Nilsen, R. M.; Vaez, A.; Fallin, M. D.; Hu, D.; Litonjua, A. A.; Fuemmeler, B. F.; Huen, K.; Kere, J.; Kull, I.; Munthe-Kaas, M. C.; Gehring, U.; Bustamante, M.; Saurel-Coubizolles, M. J.; Quraishi, B. M.; Ren, J.; Tost, J.; Gonzalez, J. R.; Peters, M. J.; Håberg, S. E.; Xu, Z.; van Meurs, J. B.; Gaunt, T. R.; Kerkhof, M.; Corpeleijn, E.; Feinberg, A. P.; Eng, C.; Baccarelli, A. A.; Benjamin Neelon, S. E.; Bradman, A.; Merid, S. K.; Bergström, A.; Herceg, Z.; Hernandez-Vargas, H.; Brunekreef, B.; Pinart, M.; Heude, B.; Ewart, S.; Yao, J.; Lemonnier, N.; Franco, O. H.; Wu, M. C.; Hofman, A.; McArdle, W.; Van der Vlies, P.; Falahi, F.; Gillman, M. W.; Barcellos, L. F.; Kumar, A.; Wickman, M.; Guerra, S.; Charles, M. A.; Holloway, J.; Auffray, C.; Tiemeier, H. W.; Smith, G. D.; Postma, D.; Hivert, M. F.; Eskenazi, B.; Vrijheid, M.; Arshad, H.; Antó, J. M.; Dehghan, A.; Karmaus, W.; Annesi-Maesano, I.; Sunyer, J.; Ghantous, A.; Pershagen, G.; Holland, N.; Murphy, S. K.; DeMeo, D. L.; Burchard, E. G.; Ladd-Acosta, C.; Snieder, H.; Nystad, W.; Koppelman, G. H.; Relton, C. L.; Jaddoe, V. W.; Wilcox, A.; Melén, E.; London, S. J., DNA Methylation in Newborns and Maternal Smoking in Pregnancy: Genome-wide Consortium Meta-analysis. Am J Hum Genet 2016, 98 (4), 680-96.
3. Horvath, S., DNA methylation age of human tissues and cell types. Genome Biol 2013, 14 (10), R115.
4. Berdasco, M.; Esteller, M., Clinical epigenetics: seizing opportunities for translation. Nat Rev Genet 2019, 20 (2), 109-127.
5. Jones, P. A.; Ohtani, H.; Chakravarthy, A.; De Carvalho, D. D., Epigenetic therapy in immune-oncology. Nat Rev Cancer 2019, 19 (3), 151-161.
6. Ahadi, S.; Zhou, W.; Schüssler-Fiorenza Rose, S. M.; Sailani, M. R.; Contrepois, K.; Avina, M.; Ashland, M.; Brunet, A.; Snyder, M., Personal aging markers and ageotypes revealed by deep longitudinal profiling. Nat Med 2020, 26 (1), 83-90.
7. Gravina, S.; Dong, X.; Yu, B.; Vijg, J., Single-cell genome-wide bisulfite sequencing uncovers extensive heterogeneity in the mouse liver methylome. Genome Biol 2016, 17 (1), 150.
Похожие статьи
Все о бисульфитной конверсии
Краткое описание и основные моменты методики
Метилирование ДНК
Знаете ли вы, что количество объятий, которые ребенок получает в детстве, может повлиять на профиль метилирования ДНК ребенка и на процесс развития? Фактически, метилирование ДНК влияет на различные аспекты повседневной жизни, о которых большинство людей даже не подозревает...
Рекомендуемые товары
D5001 Набор для бисульфитной конверсии EZ DNA Methylation Kit, 50 реакций, Zymo Research
Набор EZ DNA Methylation Kit ..
0 руб.
D5005 Набор для бисульфитной конверсии EZ DNA Methylation-Gold Kit, 50 реакций, Zymo Research
Набор EZ DNA Methylation-Gold ..
0 руб.
D5020 Набор для бисульфитной конверсии EZ DNA Methylation-Direct Kit, 50 реакций, Zymo Research
Набор EZ DNA Methylation-Direc..
0 руб.
D5030 Набор для бисульфитной конверсии ДНК короткий протокол EZ DNA Methlyation-Lightning Kit, 50 реакций, Zymo Research
Набор EZ DNA Methlyation-Light..
0 руб.
D5450 Набор для подготовки библиотек RRHP 5-hmC Library Prep Kit, 12 реакций, Zymo Research
Набор RRHP 5-hmC Library Prep ..
0 руб.
D5451 Набор для подготовки библиотек RRHP 5-hmC Library Prep Kit, 25 реакций, Zymo Research
Набор RRHP 5-hmC Library ..
0 руб.
D5455 Набор для создания библиотек Pico Methyl-Seq Library Prep Kit, 10 реакций, Zymo Research
Pico Methyl-Seq Library Prep K..
0 руб.
D5456 Набор для создания библиотек Pico Methyl-Seq Library Prep Kit, 25 реакций, Zymo Research
Pico Methyl-Seq Library Prep K..
0 руб.
D5460 Набор для создания библиотек для бисульфитного секвенирования Zymo-Seq RRBS Library Kit, 24 реакции, Zymo Research
Zymo-Seq RRBS Library Kit - эт..
0 руб.
D5461 Набор для создания библиотек для бисульфитного секвенирования Zymo-Seq RRBS Library Kit, 48 реакций, Zymo Research
Zymo-Seq RRBS Library Kit ..
0 руб.
D5013 Набор метилированной и неметилированной ДНК человека Human Methylated & Non-Methylated (WGA) DNA Set, 5 мкг/20 мкл, Zymo Research
Набор Human Methylated & N..
0 руб.
D5014 Набор метилированной/неметилированной ДНК человека Human Methylated & Non-methylated DNA Set, 5 мкг/20 мкл, Zymo Research
Набор Human Methylated & N..
0 руб.
D5011 Универсальный стандарт метилированной ДНК человека Universal Methylated Human DNA Standard, 5 мкг, Zymo Research
Universal Methylated Human DNA..
0 руб.
D5012 Универсальный стандарт метилированной ДНК мыши Universal Methylated Mouse DNA Standard, 5 мкг, Zymo Research
Universal Methylated Mouse DNA..
0 руб.
D5015 Стандарт метилированной конвертированной ДНК человека Bisulfite-Converted Universal Methylated Human DNA Standard, 1 мкг/50 мкл, Zymo Research
Bisulfite-Converted Universal ..
0 руб.
D5405 Набор стандартов ДНК 5-Methylcytosine & 5-Hydroxymethylcytosine DNA Standard Set, 1 набор, Zymo Research
Набор стандартов ДНК 5-Methylc..
0 руб.
D5016 Стандарт неметилированной ДНК E. coli Non-Methylated Genomic DNA, 5 мкг/20 мкл, Zymo Research
Стандарт неметилированной ДНК ..
0 руб.
D5017 Набор метилированной и неметилированной ДНК с праймерами Methylated & Non-methylated pUC19 DNA Set, 20 нг, Zymo Research
Набор метилированной и неметил..
0 руб.
D5209 Набор для иммунопреципитации хроматина Zymo-Spin ChIP Kit, 10 реакций, Zymo Research
Набор для иммунопреципитации х..
0 руб.
D5201 Набор для очистки и концентрирования ДНК хроматина, колонки без крышки ChIP DNA Clean & Concentrator (Uncapped Columns), 50 реакций, Zymo Research
Набор для очистки и концентрир..
0 руб.
D5206 Набор для очистки и концентрирования ДНК хроматина ZR-96 ChIP DNA Clean & Concentrator, 2x96 реакций, Zymo Research
Набор для очистки и концентрир..
0 руб.
D5465 Набор для синтеза библиотек для полногеномного бисульфитного секвенирования Zymo-Seq WGBS Library Kit, 24 реакции, Zymo Research
Набор Zymo-Seq WGBS Library Ki..
0 руб.
Теги: Эпигенетика

-228x228.png)









-80x80.jpg)