Какой метод РНК-секвенирования отдельных клеток выявляет больше генов в клетках с низким содержанием РНК?
Какой метод РНК-секвенирования отдельных клеток выявляет больше генов в клетках с низким содержанием РНК?
Если ваши исследования лежат в области РНК-секвенирования или вы являетесь в ней новичком и не знаете с чего начать, то данная статья может вам помочь.
Эксперименты в области секвенирования РНК одиночных клеток состоят из двух основных фаз: генерация кДНК и подготовка библиотеки. Набор SMART-Seq Single Cell PLUS от компании Takara BIO дает вам инструменты для выполнения обеих задач.
Данный набор сравнили с другими методиками. Компонент набора для создания кДНК (SSsc) сравнивали с популярным протоколом Smart-seq2 (SS2) и его обновленной версией Smart-seq3 (SS3). Компонент подготовки библиотеки (SSlp) сравнивался с широко используемым методом Illumina Nextera® XT. Было протестировано несколько исходных материалов: культивируемые клетки (панель A), первичные клетки (панель B) и РНК всего органа (панель C). Работа проведена с применением двух широко используемых роботов, MANTIS и mosquito HV. Полученные данные были проанализированы с помощью программного обеспечения для биоинформатики CogentAP (Takara BIO).
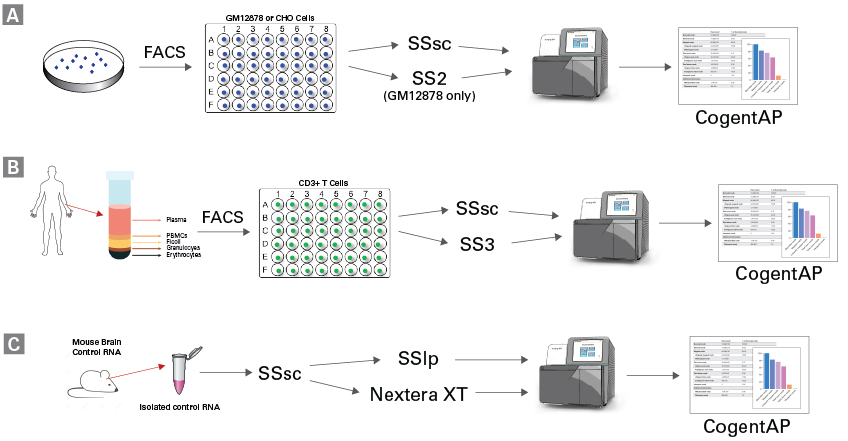
Схема рабочего процесса от Holcomb et al. J. Biomol. Tech. 32 (2021). Используется с разрешения Association of Biomolecular Resource Facilities.
Более подробную информацию о том, как комплексное решение помогает обнаружить большее количество генов при сохранении совместимости с автоматизацией и миниатюризацией рабочего процесса можно найти в статье «Benchmarking single-cell mRNA-sequencing technologies uncovers differences in sensitivity and reproducibility in cell types with low RNA content» в декабрьском выпуске журнала the Journal of Biomolecular Techniques.
Рекомендуемые товары
634470 Набор для синтеза кДНК единичных клеток SMART-Seq® Single Cell Kit, 12 реакций, Takara BIO
SMART-Seq® Single Cell Kit - э..
0 руб.
R400750 Полнокомпонентный набор для синтеза NGS библиотек единичных клеток SMART-Seq® Single Cell PLUS Kit, 48 реакций, Takara BIO
Набор SMART-Seq® Single C..
0 руб.
R400751 Полнокомпонентный набор для синтеза NGS библиотек единичных клеток SMART-Seq® Single Cell PLUS Kit, 96 реакций, Takara BIO
Набор SMART-Seq® Single C..
0 руб.
634471 Набор для синтеза кДНК единичных клеток SMART-Seq® Single Cell Kit, 48 реакций, Takara BIO
Новинка от Takara BIO SMART-Se..
0 руб.
634472 Набор для синтеза кДНК единичных клеток SMART-Seq® Single Cell Kit, 96 реакций, Takara BIO
SMART-Seq® Single Cell Kit&nbs..
0 руб.
Теги: RNA-seq, Single-cell